User:Victor Tapia
Contact Information

Víctor E . Tapia Mancilla
Email: ve.tapia.m@gmail.com
ACTUALLY SEARCHING FOR A NEW POSITION
25.02.2012
Download CV-EN
Former institutions:

Charité - Universitätsmedizin Berlin
Institut für medizinische Immunologie
AG Molekulare Bibliotheken
Dr. Rudolf Volkmer
Hessische Str. 3-4, 10115 Berlin
Tel.: +49-30-450 524267
oder +49-30-450 524317
Fax: +49-30-450 524962
E-mail: rve@charite.de
- - - - - - - - - - - - - - - - - - - - - - - -

UMR7156 CNRS-Université de Strasbourg
Equipe Cytosquelette d'actine et Trafic intracellulaire
Département de Biologie moléculaire et cellulaire
Barbara Winsor, PhD
(† 29.09.2011)
- - - - - - - - - - - - - - - - - - - - - - - -

Humboldt Universität, Berlin
Institute for Theoretical Biology
Systems Immunology
Michal Or-Guil
<html>
<a href="http://www.mendeley.com/profiles/ve-tapia"><img border="0" src="http://www.mendeley.com/embed/icon/2/red/small" alt="Bibliography manager"/></a>
</html>
last change on 28.02.2012
jet another profile
jet another other profile
Research interests
- Modularity of Protein Structure and Cellular Signal Transduction.
- Protein Folding and Neurodegenerative Deseases.
- Natural Autoimmunology and Diagnostic Assays
Scientific Projects with Principal Responsability
2010
- Complete SH3 Binding Profiles across Bacterial Strains for the Analysis of Protein Interaction Network Evolution.
- BAG3-WW Binding Profile to Elucidate the Protein's Function.
2008
- PQBP1-WW Binding Profile to Elucidate the Molecular Mechanisms of the IGH-Syndrome.
2007
- Proteosomal Degradation of Immobilized Peptides.
2006
- Quality Control of Production and Application Procedures in the Peptide-Microarray Technique.
- Characterization of Sequence Elements of A-beta Self Recognition.
2004
- Diagnosis of Immunization States with Patterns of Natural Autoimmune Reactivities.
Education
May 2010 – April 2011 |
Fellow scientific associate in “Penelope” (EU Marie Curie Research Training Network, MRTN-CT-2006- 0036076) coordinated by Dr. Luis Serrano, Centre for Genomic Regulation, Barcelona
July 2008 – April 2010 |
Fellow scientific associate in “Structure and Function of Membrane Receptors” (Collaborative Research Centre, SFB 449)
coordinated by Prof. Dr. Volker Haucke, Freie Universität, Berlin
Sept. 2003 – June 2004 |
Diploma thesis in the Systems Immunology Lab
led by Dr. M. Or-Guil at the Institute for theoretical Biology of the Humboldt Universität in collaboration with the Charité Universitätsklinikum, Berlin
Oct. 1998 – June 2004 |
Study of Biology with a Biochemistry major at the Humboldt Universität, Berlin
May 1996 – Sept. 1998 |
German as Foreign Language, German Literature and Philosophy, Epistemology, and Sociobiology as guest student at the RWTH, Aachen.
March 1979 – Dec. 1991 |
School in Viña del Mar, Chile and Philadelphia, PA, USA.
Publications

- 10 - M Spiess, E Verschueren, VE TAPIA, PM Kim, A Norgaard, C Landgraf, R Volkmer, F Hochstenbach, B Distel, B Winsor, L Serrano (2012). “EVOLUTION OF THE SH3 DOMAIN INTERACTOME ACROSS YEAST SPECIES”
- [Manuscript in preparation].
- 9 - Anna Ulbricht, Felix J. Eppler, VE TAPIA, Peter F.M. van der Ven, Padmanabhan Vakeel, Daniela Stadel, Albert Haas, Bernd Hoffmann, Paul Saftig, Christian Behrends, Dieter O. Fürst, Rudolf Volkmer, Waldemar Kolanus & Jörg Höhfeld. (2012) “CELLULAR MECHANOTRANSDUCTION RELIES ON TENSION-INDUCED AND CHAPERONE-ASSISTED AUTOPHAGY“
- [Manuscript submitted on June 29 2012]
- 8 - Volkmer, R & V Tapia. (2011) “EXPLORING PROTEIN-PROTEIN INTERACTIONS WITH SYNTHETIC PEPTIDE ARRAYS”. Mini-Reviews in Organic Chemistry 8(2): 164.
- 7 - Volkmer, R, I Kretzschmar & VE TAPIA. (2011) “Mapping receptor-ligand in-teractions with synthetic peptide arrays: Exploring the structure and function of membrane receptors”. European Journal of Cell Biology 91(4): 349.
- 6 - Tapia VE, Nicolaescu E, McDonald CB, Musi V, Oka T, Inayoshi Y, Satteson AC, Mazack V, Humbert J, Gaffney CJ, Beullens M, Schwartz CE, Landgraf C, Volkmer R, Pastore A, Farooq A, Bollen M, Sudol M. (2010) "Y65C MISSENSE MUTATION IN THE WW DOMAIN OF THE GOLABI-ITO-HALL SYNDROME PROTEIN PQBP1 AFFECTS ITS BINDING ACTIVITY AND DEREGULATES PRE-mRNA SPLICING." J Biol Chem.;285(25):19391-401. Epub 2010 Apr 21.
- This work explores the effect of a point mutation leading to X-LMR at the reductionist level of domain-peptide interactions, inferes potential affected functions, and tests inferences on pre-mRNA splicing.
- Download from JBC
- 5 - Mahrenholz CC, Tapia V, Stigler RD, Volkmer R. (2010) "A STUDY TO ASSESS THE CROSS-REACTIVITY OF CELLULOSE MEMBRANE-BOUND PEPTIDES WITH DETECTION SYSTEMS: AN ANALYSIS AT THE AMINO ACID LEVEL." J Pept Sci.;16(6):297-302.
- ever wanted to see if your favorite detection system reacts with cellulose-bound peptides? Well, chances are given that it is one of the three covered here: TAMRA-dye, luminol turn-over, and FITC
- 4 - Tapia VE & Volkmer R (2009) "EXPLORING AND PROFILING PROTEIN FUNCTION WITH PEPTIDE ARRAYS" in: Methods in Molecular Biology, Vol 570 (Eds. M Cretich & M Chiari). Humana Press. - ISBN: 978-1-60327-393-0.
- This work reviews the peptide array technology focusing on technological advancements, oriented immobilization of peptide probes, and some interesting applications. Going along the philosophy of the monography series, we give some general recomendations on methods and instruments. See the following newsletter comments by Tony Fong: Q&A: Raising the Profile of Peptide Arrays for Studying Protein Function, ProteoMonitor, August 13, 2009.
- View abstract at PubMed
- Read the newsletter by Tony Fong
- 3 - Tapia VE, Ay B, Triebus J, Wolter E, Boisguerin P, Volkmer R. EVALUATING THE COUPLING EFFICIENCY OF PHOSPHORYLATED AMINO ACIDS FOR SPOT SYNTHESIS. J Pept Sci. 2008 Dez ;14(12):1309-1314. - PMID: 18816512.
- This work was carried out to adapt "building block" approaches for the generation of phosphopeptides through resine-supported solid-phase peptide synthesis to the spot-technology, which relies on cellulose as support. It compares different coupling strategies for the incorporation of protected phoshoaminoacids into determined positions of synthetic peptides. We find that the use of EEDQ as coupling activator is the most efficient strategy in our comparison.
- view abstract at PubMed
- 2 - Tapia VE, Bongartz J, Schutkowski M, Bruni N, Weiser A, Ay B, Volkmer R, Or-Guil M. AFFINITY PROFILING USING THE PEPTIDE MICROARRAY TECHNOLOGY: A CASE STUDY. Anal Biochem. 2007 Apr 1;363(1):108-118. - PMID: 17288979.
- This work analyses the predictive potential of microarray-based binding assays. Fluorescent SI-measurements were used to fit a mass-action derived model estimating the observed affinity (equil. diss. const.).
- View abstract at PubMed
- 1 - Weiser AA, Or-Guil M, Tapia VE, Leichsenring A, Schuchhardt J, Frommel C, Volkmer-Engert R. SPOT SYNTHESIS: RELIABILITY OF ARRAY-BASED MEASUREMENT OF PEPTIDE BINDING AFFINITY. Anal Biochem. 2005 Juli 15;342(2):300-311. - PMID: 15950918.
- This work analyses the quantitative potential of measurements derived from peptide macroarray-based immunobinding assays. Chemoluminiscent signal intensity data was confronted with independent "golden stardard" measurements (surface plasmon resonance-based binding assays on a BIAcoreX maschine).
Presentations
- 6 - V.E. Tapia & R. Volkmer; Steady State Analysis of Peptide Array-based Binding Assays: A Mathematical Model for Disease Diagnostic and Affinity Profiling of Bimolecular Interactions. 9th German Peptide Symposium, 2009, Göttingen.
- 5 - Mahrenholz CC, Tapia V, Stigler R, Volkmer R; Peptides crossreacting with detection systems – Analysis at the amino acid level. 9th Peptide Symposium (2009).
- 4 - V.E. Tapia, C. Landgraf, A. Pastore, M. Sudol, R. Volkmer; Analysis of a missense mutation in the WW domain of PQBP1. FEBS Workshop 2007, Protein Modules and Networks in Health and Disease, Seefeld, Austria.
- 3 - V.E. Tapia et al; titel; 2008 Brownschweig, Germany
- 2 - V.E. Tapia et al; titel; 2007 Frankfurt, Germany
- 1 - V.E. Tapia & M. Or-Guil; Reading Natural Autoimmune Responses with Random Peptide Arrays; Workshop "Strukturbildung in Chemie und Biophysik", Salzwedel, Germany, October 2004.
Arbeitsproben
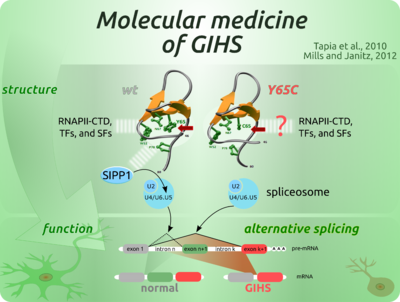
GOLABI-ITO-HALL SYNDROM (GIHS)
Das Gen fürs polyglutamine-tract binding Protein 1 (PQBP1), welches auf dem X-Chromosom lokalisiert ist, kodiert für ein 38 kDa Zellkernprotein das überwiegend im zentralen Nervensystem exprimiert wird. In der Literatur wird die Rolle des PQBP1 Proteins in der Entwicklung von auf Polyglutaminexpansion beruhenden Krankheiten wie z. B der spinozerebellären Ataxie Typ 1 beschrieben (Okazawa et al., 2002). Weiterhin sind Mutationen im PQBP1 Gen in mehreren X-Chromosom gebundenen Retardierungen (XLMR), wie dem Renpenning, Sutherland-Haan, Hamel, Porteous und Golabi-Ito-Hall Syndrom (GIHS), nachgewiesen worden (Ropers and Hamel, 2005). Dieser XLMR Syndrome assoziieren mit unterschiedlichen Mutationen des PQBP1 Gens, und dennoch teilen sie ähnliche klinische Merkmale. Die Y65C PQBP1 Mutation, die mit dem GIH Syndrom assoziiert (Lubs et al., 2006), ist einzigartig unter den bisher gemeldeten PQBP1 Mutationen. Die Läsion beeinflusst nicht die Länge des mutierten Proteins sondern betrifft eine punktuelle Position innerhalb des WW Erkennungsmodules. Mutationen, die in Erkennungsmodulen oder in ihren zugehörigen Liganden gefunden werden, beeinflussen die involvierten Proteinkomplexe und die damit verbundenen Signalwege und können zu Krankheiten führen.

Chaperon-assistierte selektive Autophagie (CASA)
CASA ist entscheidend um die Muskelaktivität unter mechanischer Spannung in Fliegen, Mäusen und Menschen (Arndt et al., 2010; Homma et al., 2006; Selcen et al., 2009) zu erhalten (Abbildung 2). Eine Beeinträchtigung der CASA verursacht schwere Muskeldystrophien und eine dilatierte Kardiomyopathie, und wurde mit der Gliedergürteldystrophie (Arimura et al., 2011; Homma et al., 2006; Sarparanta et al., 2012) in Verbindung gebracht. In Muskelzellen spielt BAG3 eine Schlüsselrolle wo es hoch exprimiert und überwiegend an der Z-Scheibe lokalisiert ist. Die Z-Scheibe ist ein Proteinkomplex und dient der Aktin-Verankerung in der quer-gestreiften Muskulatur. Für die Verankerung ist das Z-Scheiben-Protein Filamin wichtig, da es als verbindende Brücke zwischen Aktin und Integrinen in der sakroplasmatischen Membran wirkt. In der kontrahierenden Muskulatur wird das Filamin mechanisch stark beansprucht und häufig entfaltet und unterliegt dann einem Abbau durch CASA. Dieser Abbau wird duch BAG3 eingeleitet. Nach Kontraktion-induzierter Entfaltung, wird Filamin von einem Chaperon-Komplex anerkannt. Der Komplex enthält Hsc70 und HspB8 (auch als Hsp22 bekannt), die physikalisch vom CASA-induzierende Ko-Chaperon BAG3 zusammengehalten werden. Filamin wird aus der Z-Scheibe freigegeben und durch die Hsc70-assoziierte Ubiquitinligase CHIP ubiquitiniert. Es wird dann von dem autophagischen Ubiquitin Adaptor Protein p62 dem lysosomalen Verdau zugeführt, indem p62 mit Phagophorenmembranen interagiert. Der autophagische Abbau von Filamin ist eine Voraussetzung für die ordnungsmäßige Funktion von Z-Scheiben in kontrahierender, quergestreifter Muskulatur (Arndt et al., 2010).
QUELLE: Text and image to the right were adapted from the following sources:
Ulbricht, A., Eppler, F.J., Tapia, V.E., Van der Ven, P.F.M., Hampe, N., Hersch, N., Vakeel, P., Stadel, D., Haas, A., Saftig, P., Behrends, C., Fürst, D.O., Volkmer, R., Hoffmann, B., Kolanus, W., Höhfeld, J., 2013. Cellular Mechanotransduction Relies on Tension-Induced and Chaperone-Assisted Autophagy. Accepted by Current Biology
Rogon & Höhfeld, 2010. Proteostase – Chaperone als Begleiter von der Wiege bis zum Grabe. Biospektrum 4/2010; 413

INTERAKTIONSNETZWERKE
Zur Jahrhundertwende wurden einige in vivo/ex vivo Strategien entwickelt, um proteomische Interaktionsnetzwerke zu charakterisieren (Gavin et al., 2002; Ho et al., 2002; Ito et al., 2001; Uetz et al., 2000). Es zeigte sich jedoch sehr schnell, dass die berichteten Interaktome aus Saccharomyces cerevisiae nur eine mäßige Überlappung zeigen. Diese Erkenntnis fordert zwingend jegliche Interaktomstudien durch orthogonale Methoden zu validieren (Von Mering et al., 2002). Eine alternative in vitro Strategie basiert auf einer Hochdurchsatz-Protein-Präparierung im Arrayformat (Kung et al., 2009; Phizicky et al., 2003; Ptacek et al., 2005; Zhu et al., 2001). Obwohl inzwischen einige experimentelle Studien publiziert wurden, ist noch nicht klar, welcher Prozentsatz eines eukaryotischen Proteoms in gefalteter Form hergestellt und funktionsfähig auf einen festen Träger immobilisiert werden kann. Im Gegensatz zur bestehenden Unsicherheit bei Proteinarrays können hochdichte Peptidarrays effizient durch eine Spot Synthese präpariert werden (Frank, 2002; Hilpert et al., 2007; Winkler et al., 2011). Der Informationsverlust durch Verzicht auf den Proteinkontext wird durch die Tatsache relativiert, dass erkannte kurze lineäre motiven in nativen unstrukturierten Regionen der Proteinarchitektur exponiert werden (Dinkel et al., 2011; Fuxreiter et al., 2007). Der Vorteil des Peptidarray-Formats erschließt sich für jene Protein-Protein-Wechselwirkungen, in denen einer der Bindungspartner die Komplexbildung durch Andocken an einen kurzen linearen Sequenzabschnitt seines Partnerproteins auslöst . In der Tat wird eine beträchtliche Menge von Protein-Protein-Interaktionen durch Familien von Erkennungsdomänen vermittelt (WW, SH3, SH2, PDZ usw.), die kurze lineare Peptide in ihrer Bindungstaschen aufnehmen (Hunter and Pawson, 2012; Pawson and Linding, 2008, 2005; Pawson and Nash, 2000). Eine vergleichende Analyse der Evolution von SH3-Netzwerke wurde zusammen mit Kooperationspartnern, wie in 3 skizziert, durchgeführt (Verschueren et al., n.d.).
Useful links
Who's visiting
start on 18 Mar 2010
<html> <a href="http://www2.clustrmaps.com/counter/maps.php?url=http://openwetware.org/wiki/User:Victor_Tapia" id="clustrMapsLink"><img src="http://www2.clustrmaps.com/counter/index2.php?url=http://openwetware.org/wiki/User:Victor_Tapia" style="border:0px;" alt="Locations of visitors to this page" title="Locations of visitors to this page" id="clustrMapsImg" onerror="this.onerror=null; this.src='http://clustrmaps.com/images/clustrmaps-back-soon.jpg'; document.getElementById('clustrMapsLink').href='http://clustrmaps.com';" /> </a> </html>